Articulo original publicado por la Delegación del CSIC en la Comunitat Valenciana el 27 .09.2021
Año y medio después de la irrupción de la pandemia de COVID-19 se ha publicado en la revista Nature Genetics el estudio científico más completo sobre las variantes del coronavirus que circularon por España durante la ‘primera ola’. En él han colaborado científicos del Centro de Investigación en Sanidad Animal (CISA) y del Departamento de Mejora Genética Animal del INIA-CSIC, como integrantes de SeqCOVID, consorcio del que forma parte el INIA-CSIC y que engloba a más de 50 instituciones de investigación y sanitarias del país coordinadas por el Instituto de Biomedicina de Valencia (IBV-CSIC) junto con la Universitat de València y la Fundación para el Fomento de la Investigación Sanitaria y Biomédica de la Comunitat Valenciana (Fisabio).
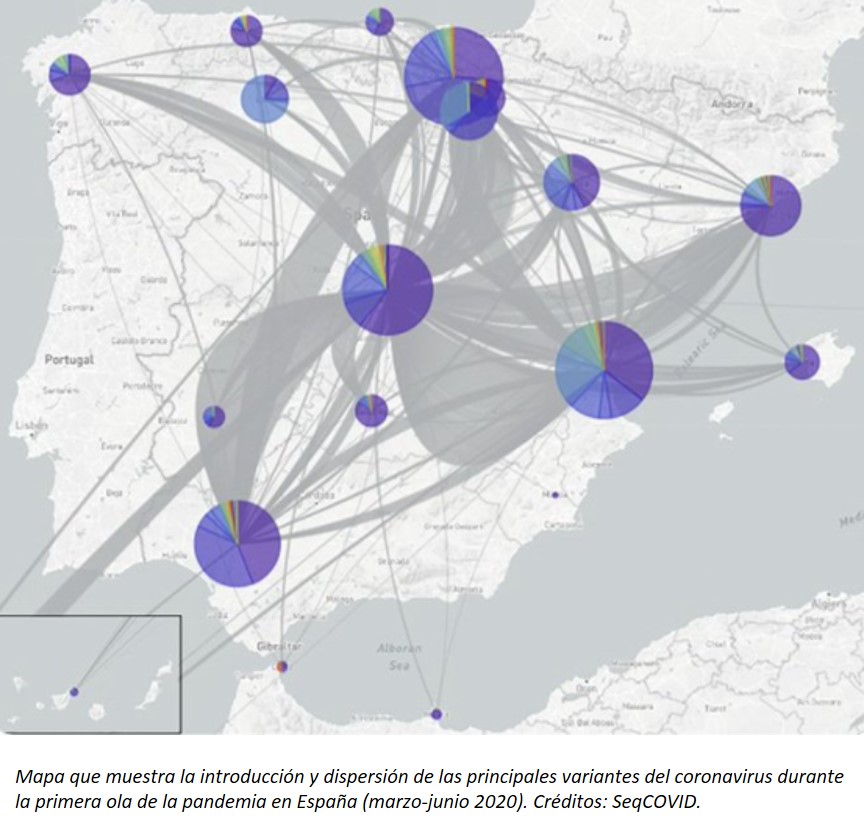
Para la realización del estudio, los investigadores del consorcio SeqCOVID secuenciaron 2.500 muestras procedentes de pacientes diagnosticados en España durante la primera ola de la pandemia, suponiendo un 1% de todos los casos diagnosticados entre marzo y junio de 2020.
En concreto, los investigadores del CISA y del Departamento de Mejora Genética del INIA-CSIC han aportado las secuencias de SARS-CoV-2 obtenidas de muestras recibidas durante el dispositivo de análisis masivo mediante PCR implementado en el INIA-CISA/CSIC al inicio del estado de alarma, que se organizó junto con el Ayuntamiento de Madrid para la vigilancia de sus servicios esenciales.
El estudio identifica 9 variantes del virus (denominadas SEC, del inglés Spanish Epidemic Clades), que fueron las que dominaron esta primera ola en España. Dos de ellas (SEC7 y SEC8) fueron las primeras detectadas en el país y las predominantes durante ese periodo, y se asocian al menos a dos eventos de superdispersión conocidos: el partido Atalanta-Valencia de la Champions League y un funeral de Vitoria, aunque se identifican focos tempranos en otras partes del país. No hubo una única introducción del virus en España sino múltiples entradas independientes (al menos 500), desde distintos orígenes internacionales. Estas se dieron principalmente durante febrero y marzo de 2020, antes de la implementación de las medidas de control.
“La mayoría de infecciones de la primera ola antes del confinamiento en España fueron provocadas por cepas del linaje A del coronavirus. Estas eran abundantes en los países asiáticos en aquel momento, pero tenían menos presencia en el resto de países europeos, donde dominaban cepas de linaje B. Esto no quiere decir que las introducciones de SARS-CoV-2 en nuestro país fueran mayoritariamente asiáticas. En realidad, vemos que la mayoría de introducciones provienen de países europeos, pero las cepas de linaje A se establecieron antes y, gracias a eventos de superdispersión, se expandieron en nuestro país rápidamente”, explica Álvaro Chiner, uno de los investigadores del CSIC en el IBV de Valencia que participó en el estudio (los investigadores observaron un patrón similar para la variante que dominó en la segunda ola, según datos que se acaban de publicar en la revista Nature).
El trabajo también cuantifica la efectividad de las medidas implementadas para el control del virus. Durante la primera ola todas las variantes identificadas redujeron su prevalencia y transmisión significativamente a partir del estado de alarma. Prácticamente desaparecieron al final de la primera ola, siendo reemplazadas por nuevas variantes que surgieron en el verano, cuando se relajaron las medidas de control.
“El confinamiento fue altamente efectivo para parar la transmisión del virus. No sólo para las variantes dominantes SEC7 y SEC8, sino para todas las que circulaban en aquel momento, incluyendo aquellas que contenían la mutación del gen S llamada D614G, que fue la primera que demostró incrementar la transmisibilidad del virus”, remarca Mariana López, investigadora del CSIC en el IBV y coautora del estudio.
“Algo similar estamos viendo a través de las olas. Estamos detectando variantes más transmisibles, pero su impacto se puede controlar con las medidas de control adecuadas. Allá donde esas medidas no han existido o son más relajadas las variantes han agravado los rebrotes. Ocurrió en el Reino Unido con la variante Alpha y está ocurriendo en España con Delta” indica Iñaki Comas, investigador del CSIC en el IBV coordinador de SeqCOVID.
El estudio ha puesto de manifiesto la necesidad de incorporar la epidemiología genómica como una herramienta más de salud pública para rastrear el virus e identificar las variantes de mayor impacto. “La vigilancia activa de mutaciones virales debe continuar para poder evaluar la amenaza de nuevas variantes con riesgos potenciales para controlar la epidemia. Ese es uno de los objetivos que nos hemos puesto dentro de la Plataforma Temática Interdisciplinar de Salud Global del CSIC” comenta Mireia Coscollá, investigadora de la Universitat de València en el Instituto de Biología Integrativa de Sistemas (I2SysBio, CSIC-UV) y una de las coordinadoras del estudio.
En línea con lo anterior, el consorcio SeqCOVID ha contribuido a la creación de una Red de Vigilancia Genómica Nacional, dirigida por el Centro de Coordinación de Alertas y Emergencias Sanitarias (CCAES) y coordinada por el Instituto de Salud Carlos III (ISCIII), que está totalmente integrada dentro de las labores asistenciales de los hospitales.
Más información:
López, M.G., Chiner-Oms, Á., García de Viedma, D. et al., The first wave of the COVID-19 epidemic in Spain was associated with early introductions and fast spread of a dominating genetic variant, Nature Genetics (2021). DOI: https://doi.org/10.1038/s41588-021-00936-6
Hodcroft, E.B., Zuber, M., Nadeau, S. et al., Spread of a SARS-CoV-2 variant through Europe in the summer of 2020, Nature (2021). DOI: https://doi.org/10.1038/s41586-021-03677-y
